皆さんこんにちは。
@tereka114です。
昨年の年末までKaggleで開催された「Sartorius - Cell Instance Segmentation」に取り組み、4位を獲得しました。
本記事では、簡単にそのサマリを掲載します。

コンペティション概要
画像の中から、脳の試験細胞を見分けるコンペティションです。
具体的には細胞を個別にSegmentationする所謂、Instance Segmentationを行うコンペティションです。
Detectionは物体の領域に矩形を付けるものになりますが、Instance SegmentationはDetectionした上で、矩形の中の前景を抽出します。
投薬の反応によって、神経病(アルツハイマーなど)の病気に対する創薬に役立てるようですが、この確認に非常に手間がかかっているそうです。
そのため、セルのInstance Segmentationを試み、反応を自動的に判断するのに役立つコンペが開催されました。
細胞のサンプルは次の通りです。
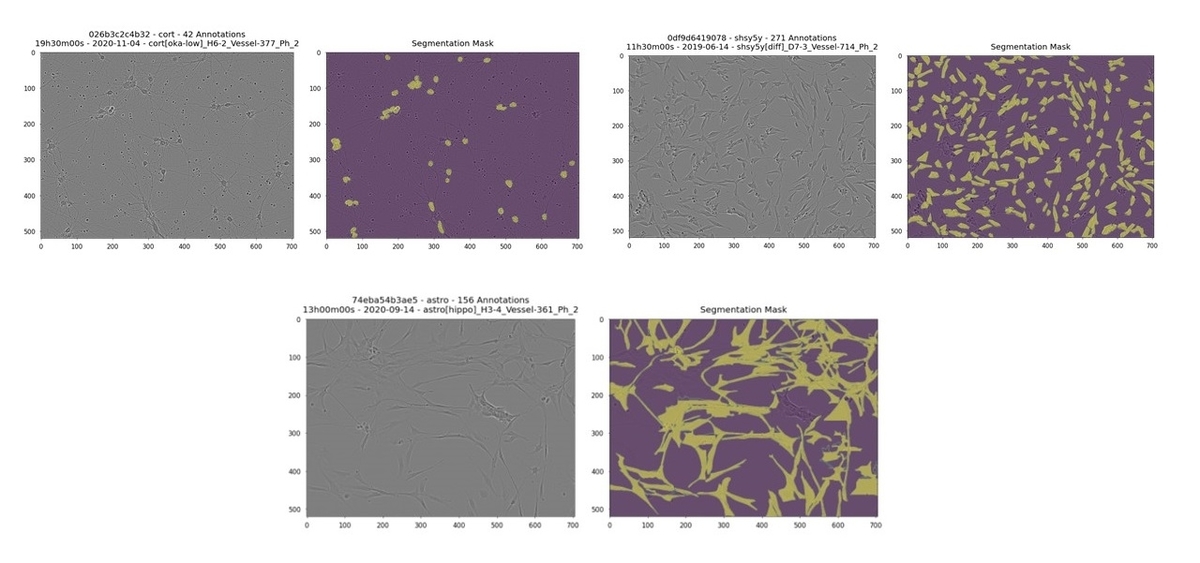
※本可視化には次のNotebookの画像を利用させていただいています。
Sartorius Cell Instance Segmentation - EDA | Kaggle
このコンペティションに参加した理由は次の通りです。
- 最近、Instance Segmentationのコンペティションは開催されておらず、興味があったこと。
- 外部データや正確なラベルがないSemi-Supervised Learning用のデータセットが配布されており、データに工夫の余地が多くあり、面白そうだと思ったこと
今回のコンペティションで難しいポイントは次の通りです。
1. 細胞の数
Detection/Instance Segmentationの論文の多くで利用されているCOCOのデータセットの1画像中最も多いオブジェクト数は92です。
しかし、今回の細胞データでは細胞の種類(astro, shsy5h, cort)によって異なりますが、最大700をも超えるオブジェクト数が存在します。
そのため、COCOのデータで最適化されたモデルでは不十分で、多くの物体を検出するには、パラメータの一部を修正する必要がありました。
2. データセット
このコンペでは、Kaggleで外部データであるLive CellとSemi-supervised learningで利用するデータセットが用意されていることが特徴的です。
LiveCellはコンペティションで提供されているものがshsy5hと呼ばれる細胞の種類以外は別のものではありますが、Segmentationのアノテーションがされています。
また、Semi-Supervised learningの画像には、細胞の種類は提供されていますが、マスク情報は存在しないため、そのまま利用できません。
ソリューション
全体の流れ
チーム全体の詳細はこちらに記載しています。
www.kaggle.com
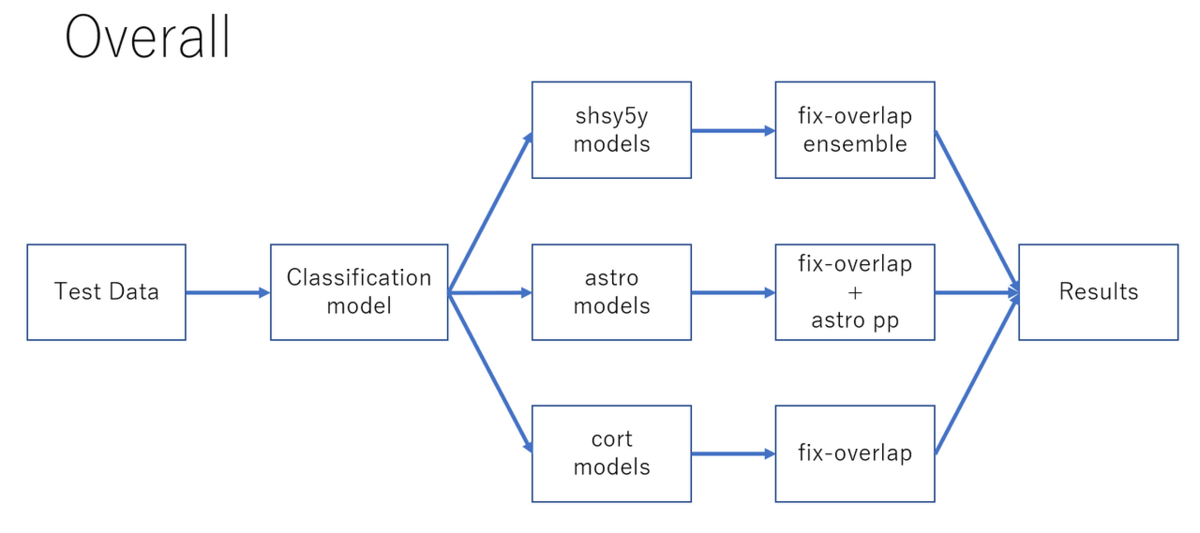
全体の流れは次の通りです。
- Instance Segmentitonで得られた結果に基づいて、細胞全体の画像を分類する。(単純に分類されたセルの多いものを採用)
- 分類した細胞ごとに別々のInstance Segmentationのモデルを用いる。
- 細胞ごとにアンサンブルやSegmentation Modelを用いたPost Process、Fix Overlap処理を行う。
※図中のshsy5y, astro, cortは細胞名由来の名称
Fix Overlapは、本コンペティションでは、提出形式としてセルのマスクの重複を許容していないため、その対応の処理です。
(細胞Aと細胞Bで同じPixelを指定できないので、片方にする必要がある。)
また、細胞に関して後処理に違いがあるのは、各処理を適用した際にCV/LBを見て精度が上がったものと下がったものがあり、その違いが後処理の内容に反映されています。
モデル作成
Classification/セルのマスク取得にはInstance Segmentationのモデルを作成しました。
具体的には次の手順を踏んでいます。
- モデルはCBNetV2 + CascadeRCNNやResNeXt101+Hybrid Task Cascadeを利用
- それぞれの細胞ごとにモデルとハイパーパラメータ群をチューニング
- Semi-Supervised Learningのデータに学習済のモデルでラベルを付与(後述)
- 事前学習として、Live Cellを学習した後、Semi Supervised Learningのデータで再学習(後述)
CBNetV2 + CascadeMaskRCNN(CRNN)とResNeXt101 + Hybrid Task Cascade(HTC)を用いたのは、最終的に精度が良かったためです。
他にもDetectors + HTC、ResNet50 + HTC、さらにはSwin Transformer + CRNNなど様々なモデルを試していましたが、アンサンブルなどに利用しても精度向上に貢献しませんでした。
チーム全体の実装はDetection/Instance Segmentationのフレームワークmmdetectionを利用しました。
設定のみで様々なアルゴリズムの利用ができる&学習済モデルが多いので、個人的にもYoloX/YoloV5を利用しないときにはmmdetectionをよく利用します。
今回は、COCOでSoTAを達成した物体検出の方式であるCBNetV2を利用したかったため、まだ実装されていない既存のmmdetectionに対してCBNetV2が追加されたリポジトリを用いました。
ハイパーパラメータ群のチューニング
mmdetectionは様々なモデルのconfigと学習済モデルが配布されています。
SoTAの計測がCOCOである論文がほとんどであるため、mmdetectionの物体検出パラメータはCOCOに最適化されています。
しかし、COCOとはデータセットの傾向が大きく異なるため、ハイパーパラメータを今回のデータセットに向けて大きく変更すると精度が向上しました。
具体的には、次のパラメータを変更して実行していました。
COCOと比較して、オブジェクト数が多いので、NMSでフィルタリングする数をCOCOよりも緩めると、精度向上します。
rpn_proposal.nms_pre=4000, rpn_proposal.nms_post=4000, rpn_proposal.max_per_img=4000 anchor_generator.ratios=[0.25, 0.5, 1.0, 2.0, 4.0]
外部&追加データの検討
先にも簡単に書きましたが、外部データであるLiveCellと追加データとしてSemi-Supervised Learningを行うためのデータが配布されていました。
これらのデータを有効に利用すると精度向上する可能性がありました。
Semi-Supervised Learningのデータは細胞の種類が記載されたCSVがありますが、マスクのアノテーションは存在していません。
学習にこのデータを利用する方法としていくつかありますが、チームでは学習モデルから予測した結果であるPseudo Labelingをすることで、学習に使えるようにしました。
1. LiveCell + Trainingデータを用いて学習したモデルを利用して、Semi-Supervised Learningを推論し、アノテーションする。
2. LiveCellで事前学習したモデルに対して、Semi-Supervised Learningのアノテーションした画像を学習する。
3. 最後にTrainingのデータでFinetuneする。
2で作成されたラベルはノイズが含まれているので、その影響を修正する役割として3を実行しました。
Semi-Supervised LearningとTrainingのデータを同時に学習させた場合と比較して、2,3のステップを踏むほうが精度が向上します。
最後に
Instance Segmentationは検討する内容も多く、コンペ終了後に他チームの共有されたソリューションを振り返ると様々な問題の解き方がありました。
これをうまく業務にも応用していきたいと考えています。
Acroquest Technologyでは、キャリア採用を行っています。
- ディープラーニング等を使った自然言語/画像/音声/動画解析の研究開発
- Elasticsearch等を使ったデータ収集/分析/可視化
- マイクロサービス、DevOps、最新のOSSを利用する開発プロジェクト
- 書籍・雑誌等の執筆や、社内外での技術の発信・共有によるエンジニアとしての成長
少しでも上記に興味を持たれた方は、是非以下のページをご覧ください。Kaggle Grandmasterと話したいエンジニアWanted! - Acroquest Technology株式会社のデータサイエンティストの求人 - Wantedlywww.wantedly.com